دوره NGS منتشر شد …

I-TASSER (Iterative Threading ASSEmbly Refinement) یکی از پیشرفتهترین و کارآمدترین سرورهای پیشبینی ساختار پروتئین در دنیای بیوانفورماتیک است. این نرمافزار که توسط گروه تحقیقاتی پروفسور یانگ ژانگ در دانشگاه میشیگان توسعه یافته، توانسته با ترکیبی هوشمندانه از روشهای threading، مدلسازی ab initio و بهینهسازی ساختاری، دقت قابل توجهی در پیشبینی ساختار سهبعدی پروتئینها به دست آورد. در مسابقات CASP (Critical Assessment of protein Structure Prediction) که مهمترین رقابت ارزیابی روشهای پیشبینی ساختار پروتئین است، I-TASSER (سرواژه معادل فارسی: آیتاسر) به طور مداوم در صدر برترینها قرار داشته و به عنوان استاندارد طلایی در این حوزه شناخته میشود.
توسعه I-TASSER در اوایل دهه 2000 میلادی آغاز شد و از آن زمان تاکنون، این نرمافزار مسیر تکاملی قابل توجهی را طی کرده است. نسخه اولیه این نرمافزار با نام TASSER شناخته میشد که بعدها با اضافه شدن قابلیتهای تکراری و بهبود الگوریتمها به I-TASSER تغییر نام داد. در طول سالهای گذشته، این نرمافزار با افزودن قابلیتهای جدید مانند پیشبینی عملکرد پروتئین (COACH)، آنالیز جایگاههای اتصال لیگاند و پیشبینی ساختار پروتئینهای غشایی (GPCR-I-TASSER) تکامل یافته است. امروزه، سرور I-TASSER یکی از پراستفادهترین ابزارهای آنلاین در جامعه محققان بیوشیمی و بیولوژی ساختاری است که ماهانه هزاران درخواست از سراسر دنیا را پردازش میکند.
در اکوسیستم متنوع ابزارهای پیشبینی ساختار پروتئین، I-TASSER جایگاه ویژهای دارد. این نرمافزار با ارائه یک رویکرد ترکیبی که از مزایای روشهای مبتنی بر بندکشی و ab initio بهره میبرد، توانسته است کارایی بالایی حتی در پروتئینهای چالشبرانگیز با ساختارهای جدید یا همولوگهای دور نشان دهد. برخلاف برخی ابزارهای محدود که تنها به پیشبینی ساختار اکتفا میکنند، آیتاسر یک پلتفرم جامع است که از پیشبینی ساختار گرفته تا آنالیز عملکردی، شناسایی جایگاههای اتصال و حتی پیشنهاد مکانیسمهای مولکولی را پوشش میدهد. این ویژگی آیتاسر را به انتخاب اول بسیاری از محققان در پروژههای مرتبط با بیولوژی ساختاری، طراحی دارو و مطالعات تکاملی تبدیل کرده است.
ابتدا از طریق لینک زیر به سرور آیتاسر وارد میشویم. مطابق با تصویر زیر توالی پروتئینی را به همراه نام کاربری و رمز عبور وارد مینماییم.
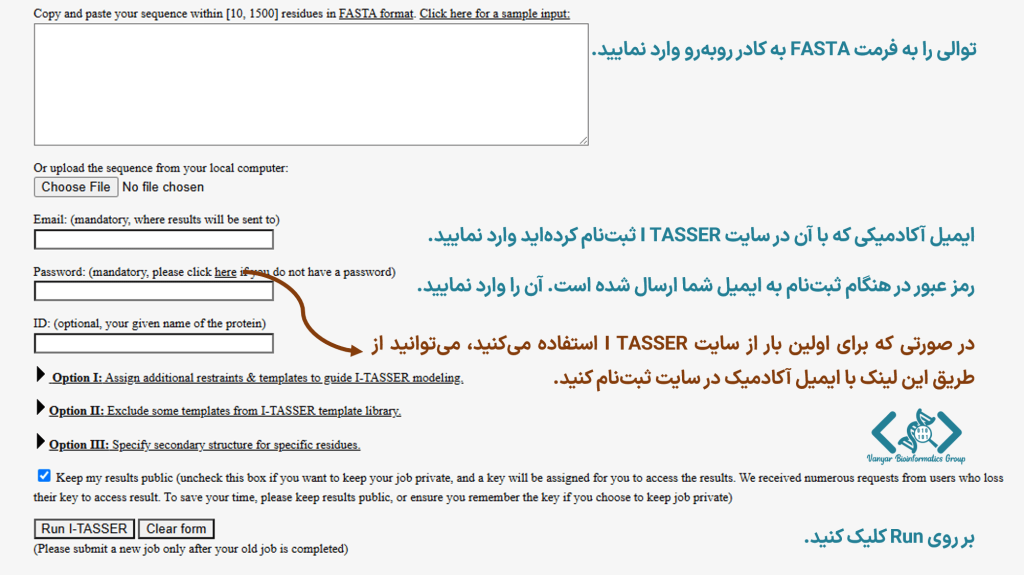
زمان اجرا و محاسبه مدلسازی در سرور آیتاسر نسبت به سایر ابزارهای بیوانفورماتیکی کمی طولانی میباشد، پس از اتمام مدلسازی نتایج از طریق ایمیل برای شما ارسال خواهد شد.
در بخش بالایی صفحه نتایج شما میتوانید تمامی نتایج را به صورت فشرده دانلود نمایید.
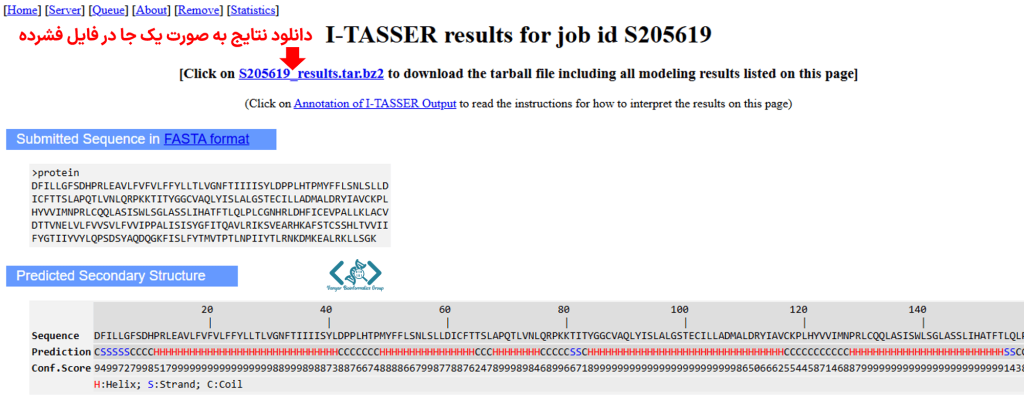
در بخش مدلهای برتر شما میتوانید بهترین مدل با امتیاز C-score برتر را در قالب فایل PDB دانلود نمایید.

در قلب الگوریتم I-TASSER، روش threading (بندکشی) قرار دارد که اساس شناسایی الگوهای ساختاری مناسب از میان پروتئینهای با ساختار شناختهشده است. در این روش، توالی هدف روی ساختارهای موجود در کتابخانه الگو “پوشیده” میشود و بر اساس سازگاری توالی-ساختار و نمرات انرژی، بهترینها انتخاب میشوند. آیتاسر از ابزار قدرتمند LOMETS (Local Meta-Threading Server) استفاده میکند که ترکیبی از چندین روش مختلف threading است. این رویکرد ترکیبی قدرت تشخیص را افزایش داده و اجازه میدهد حتی پروتئینهای آنالوگ نیز شناسایی شوند. الگوهای انتخاب شده سپس به قطعات کوچکتر شکسته شده و در مراحل بعدی بازسازی استفاده میشوند.
پیشنهاد: در دوره بیوانفورماتیک پروتئین سرور I TASSER به همراه تمامی ابزارهای پرکاربرد در حوزه بیوانفورماتیک پروتئینها آموزش داده شده.

پس از شناسایی الگوهای مناسب، I-TASSER وارد مرحله بازسازی ساختار میشود. در این مرحله، از یک فرآیند شبیهسازی مونت کارلو تحت کنترل یک میدان نیروی ترکیبی استفاده میشود. این میدان نیرو شامل اطلاعات حاصل از پیشبینیهای ساختار ثانویه، محدودیتهای فاصلهای استخراج شده از الگوها، و پتانسیلهای آماری مبتنی بر دانش است. شبیهسازی مونت کارلو به الگوریتم اجازه میدهد فضای گستردهای از کنفورماسیونهای ممکن را جستجو کند و از دامهای انرژی محلی فرار نماید. این فرآیند به صورت تکراری انجام میشود و در هر تکرار، ساختارهای قبلی به عنوان قیدهای جدید به کار میروند. نتیجه این شبیهسازیها، مجموعهای از حالتهای کنفورماسیونی است که در مرحله بعد خوشهبندی میشوند.
یکی از مهمترین ویژگیهای I-TASSER، سیستم امتیازدهی دقیق آن برای ارزیابی کیفیت مدلهای پیشبینی شده است. این نرمافزار از چندین معیار برای سنجش اعتبار مدلها استفاده میکند که مهمترین آنها C-score (نمره اطمینان) است. C-score یک نمره ترکیبی است که بر اساس کیفیت الگوهای threading، تراکم خوشهبندی و معیارهای انرژی محاسبه میشود و معمولاً در محدوده -5 تا 2 قرار دارد، که نمرات بالاتر نشاندهنده اطمینان بیشتر به مدل است. علاوه بر این، آیتاسر مقادیر تخمینی TM-score (معیار شباهت توپولوژیکی) و RMSD (جذر میانگین مربعات انحراف) را نیز ارائه میدهد که میزان نزدیکی ساختار پیشبینی شده به ساختار واقعی را نشان میدهند. مدلهای با TM-score بالاتر از 0.5 عموماً دارای توپولوژی درست هستند، در حالی که مدلهای با RMSD کمتر از 4 آنگستروم بسیار دقیق محسوب میشوند.
یکی از برجستهترین کاربردهای I-TASSER در پیشبینی ساختار پروتئینهایی است که همولوگ شناختهشدهای در پایگاههای داده ساختاری ندارند. این مورد خصوصاً در مطالعه پروتئینهای بیماریزا، پروتئینهای جدید کشف شده در ژنومهای تازه توالییابی شده، و پروتئینهای منحصر به فرد در مسیرهای متابولیکی خاص اهمیت مییابد. در چنین مواردی، آیتاسر با بهرهگیری از قطعات ساختاری شناسایی شده در پروتئینهای دیگر و الگوریتمهای بازسازی ab initio، میتواند مدلهای قابل اعتمادی ارائه دهد. تحقیقات نشان داده است که حتی در پروتئینهای با کمتر از 30% شباهت توالی به ساختارهای شناختهشده، آیتاسر توانسته مدلهایی با TM-score بالاتر از 0.5 تولید کند که برای بسیاری از کاربردهای تحقیقاتی کافی است.
در حوزه طراحی دارو، ساختارهای پروتئینی پیشبینی شده توسط آیتاسر نقش کلیدی در فرآیند غربالگری مجازی و داکینگ مولکولی ایفا میکنند. بسیاری از اهداف دارویی جدید، ساختار کریستالوگرافی شناختهشدهای ندارند و این جاست که آیتاسر به کمک محققان میآید. مدلهای با کیفیت بالای تولید شده توسط این نرمافزار میتوانند به عنوان ورودی برای نرمافزارهای داکینگ مانند AutoDock، GOLD یا Glide استفاده شوند. علاوه بر این، پیشبینیهای جایگاه اتصال لیگاند که توسط ماژول COACH در I-TASSER ارائه میشود، میتواند فرآیند طراحی دارو را هدفمندتر کند. محققان با استفاده از این اطلاعات میتوانند روی مناطق خاصی از پروتئین هدف تمرکز کنند که احتمال اتصال لیگاندها در آنها بیشتر است، و بدین ترتیب کارایی فرآیند غربالگری مجازی را به طور چشمگیری افزایش دهند.
I-TASSER فراتر از پیشبینی ساختار، ابزارهایی برای پیشبینی عملکرد پروتئین بر اساس شباهت ساختاری نیز ارائه میدهد. ماژول COFACTOR در این نرمافزار، مدل ساختاری پیشبینی شده را با پایگاههای داده عملکردی مانند Gene Ontology، مقایسه میکند و عملکردهای مولکولی، فرآیندهای بیولوژیکی و اجزای سلولی مرتبط را شناسایی مینماید. این قابلیت به خصوص در مطالعه پروتئینهای با عملکرد ناشناخته یا پروتئینهای پیشبینی شده حاصل از پروژههای ژنومیکس ارزشمند است. محققان با استفاده از این اطلاعات میتوانند فرضیههای جدیدی درباره نقش پروتئینهای مورد مطالعه خود ایجاد کرده و آزمایشهای تجربی هدفمندتری طراحی کنند. مطالعات متعددی نشان دادهاند که پیشبینیهای عملکردی آیتاسر در بسیاری موارد با نتایج تجربی مطابقت قابل قبولی دارند.
برای استفاده بهینه از سرور I-TASSER، آمادهسازی صحیح دادههای ورودی از اهمیت بسزایی برخوردار است. حداقل ورودی مورد نیاز، توالی اسیدهای آمینه پروتئین هدف در فرمت FASTA است. برای بهبود نتایج، کاربران میتوانند اطلاعات اضافی نیز ارائه دهند. به عنوان مثال، اگر ساختار پروتئینهای همولوگ خاصی را میشناسید که میخواهید در فرآیند مدلسازی استفاده شوند، میتوانید PDB ID آنها را مشخص کنید. همچنین، امکان ارائه قیدهای فاصلهای (distance constraints) یا اطلاعات اتصال دیسولفید وجود دارد که میتواند دقت پیشبینی را افزایش دهد. برای پروتئینهای بزرگ (بیش از 1500 اسید آمینه)، توصیه میشود که ابتدا آنها را به دامنههای کوچکتر تقسیم کنید، زیرا هم دقت پیشبینی بهتر خواهد بود و هم زمان محاسبات کاهش مییابد.
I-TASSER امکانات متنوعی برای تنظیم پارامترهای پیشبینی ارائه میدهد که کاربران پیشرفته میتوانند از آنها بهره ببرند. یکی از مهمترین این گزینهها، تعیین محدوده پیشبینی ساختار ثانویه است. اگر اطلاعات قابل اعتمادی از آزمایشهای CD یا NMR درباره ساختار ثانویه بخشهایی از پروتئین دارید، میتوانید این اطلاعات را به عنوان قید وارد کنید. همچنین، امکان مشخص کردن قطعاتی از پروتئین که باید از الگوی خاصی پیروی کنند وجود دارد. در حالت پیشفرض، آیتاسر از همه ساختارهای موجود در PDB به عنوان الگوهای بالقوه استفاده میکند، اما اگر میخواهید مجموعه الگوها را محدود کنید (مثلاً فقط ساختارهای انسانی یا باکتریایی)، این امکان هم فراهم است. برای پروتئینهای غشایی، توصیه میشود از نسخه اختصاصی GPCR-I-TASSER استفاده شود که الگوریتمهای بهینهشده برای این نوع پروتئینها دارد.
پس از تکمیل فرآیند پیشبینی، I-TASSER نتایج متنوعی ارائه میدهد که تفسیر صحیح آنها برای استفاده بهینه ضروری است. خروجی اصلی شامل پنج مدل با بالاترین امتیاز است که با نامهای Model 1 تا Model 5 مشخص میشوند. برای هر مدل، مقدار C-score که نشاندهنده اعتبار کلی مدل است، محاسبه میشود. به طور کلی، مدلهای با C-score بالاتر از -1.5 قابل اعتماد در نظر گرفته میشوند. علاوه بر این، آیتاسر تخمینی از TM-score و RMSD را نیز ارائه میدهد که نشان میدهد مدل پیشبینی شده چقدر به ساختار واقعی نزدیک خواهد بود. در بخش آنالیز عملکردی، پیشبینیهای مربوط به جایگاههای اتصال لیگاند، سایتهای فعال آنزیمی و عبارات Gene Ontology همراه با نمرات اطمینان آنها ارائه میشود. همچنین فایلهای مربوط به الگوهای threading استفاده شده و ماتریس تماس پروتئین نیز در دسترس است که میتواند برای تحلیلهای پیشرفتهتر مورد استفاده قرار گیرد.
با ظهور AlphaFold 2 از شرکت DeepMind در سال 2020، انقلابی در دنیای پیشبینی ساختار پروتئین رخ داد. این ابزار مبتنی بر یادگیری عمیق توانست با دقتی نزدیک به روشهای تجربی، ساختار پروتئینها را پیشبینی کند. در مقایسه با I-TASSER، AlphaFold 2 در پروتئینهای با ساختار تک دامنه عملکرد بهتری دارد و معمولاً RMSD پایینتری ارائه میدهد. با این حال، I-TASSER هنوز در برخی زمینهها مزایایی دارد. اول اینکه آیتاسر یک چارچوب جامعتر برای تحلیل عملکردی پروتئینها ارائه میدهد که شامل پیشبینی جایگاههای اتصال، پتانسیل برهمکنش با سایر مولکولها و عبارات Gene Ontology است. دوم، در پروتئینهای چند دامنه و پیچیده، آیتاسر با رویکرد ترکیبی خود گاهی نتایج بهتری ارائه میدهد. سوم، آیتاسر انعطافپذیری بیشتری در پذیرش قیدهای کاربر-تعریف (user-defined constraints) دارد که برای مواردی که اطلاعات تجربی جزئی در دسترس است، بسیار مفید است.
آیتاسر علیرغم قدرت و محبوبیت قابل توجه، محدودیتهایی نیز دارد که آگاهی از آنها برای استفاده صحیح ضروری است. از مزایای اصلی این نرمافزار میتوان به جامعیت آن (پیشبینی ساختار و عملکرد)، انعطافپذیری در پذیرش قیدهای مختلف، توانایی مدلسازی پروتئینهای بدون همولوگ نزدیک، و امتیازدهی دقیق به مدلها اشاره کرد. از طرف دیگر، محدودیتهای آیتاسر شامل زمان محاسباتی طولانی (گاهی تا چند روز برای پروتئینهای بزرگ)، چالش در مدلسازی صحیح حلقههای بسیار بزرگ و متغیر، و کارایی نسبتاً پایینتر در مقایسه با AlphaFold 2 برای پروتئینهایی که همولوگ نزدیک دارند، میشود. همچنین، آیتاسر در مدلسازی کمپلکسهای پروتئین-پروتئین بزرگ محدودیت دارد، هرچند نسخههای تخصصیتر مانند SPRING و MM-I-TASSER برای رفع این محدودیت توسعه یافتهاند.
در سالهای اخیر، گروه توسعهدهنده آیتاسر پیشرفتهای قابل توجهی در بهبود الگوریتمها و گسترش قابلیتهای این نرمافزار داشتهاند. یکی از مهمترین این پیشرفتها، توسعه C-I-TASSER (Contact-guided I-TASSER) است که از اطلاعات پیشبینی تماسهای پروتئینی با استفاده از روشهای یادگیری عمیق بهره میبرد. این رویکرد موجب بهبود چشمگیر دقت در پروتئینهای چالشبرانگیز شده است. همچنین، QuarkI-TASSER که ترکیبی از الگوریتمهای مدلسازی ab initio Quark و I-TASSER است، برای پروتئینهایی که هیچ قالب ساختاری مناسبی ندارند، توسعه یافته است. برای پروتئینهای غشایی، GPCR-I-TASSER و GPCR-RaptorX با الگوریتمهای تخصصی ارائه شدهاند.
یکی از هیجانانگیزترین روندهای اخیر در توسعه I-TASSER، تلفیق آن با روشهای یادگیری عمیق است. با توجه به موفقیت چشمگیر AlphaFold 2، گروه توسعهدهنده آیتاسر نیز شروع به ادغام تکنیکهای یادگیری عمیق در چارچوب موجود خود کردهاند. به عنوان مثال، نسخه جدید D-I-TASSER از شبکههای عصبی عمیق برای پیشبینی ماتریسهای تماس و زوایای دیهدرال استفاده میکند که سپس به عنوان قید در فرآیند مدلسازی آیتاسر به کار میروند. نتایج اولیه نشان میدهد که این رویکرد ترکیبی میتواند مزایای هر دو روش را با هم داشته باشد: دقت بالای مدلهای مبتنی بر یادگیری عمیق و توانایی تحلیل جامع ساختار-عملکرد I-TASSER. در آینده نزدیک، انتظار میرود نسخههای جدیدتری از I-TASSER ارائه شوند که به طور کاملتری از قدرت یادگیری عمیق برای بهبود همه جنبههای پیشبینی، از مدلسازی ساختار گرفته تا تحلیل عملکردی و شناسایی برهمکنشها، بهره ببرند.
I-TASSER به عنوان یک پلتفرم جامع برای پیشبینی ساختار و عملکرد پروتئین، نقش مهمی در پیشرفت مطالعات بیوانفورماتیک و بیولوژی ساختاری ایفا کرده است. قدرت این نرمافزار در ترکیب هوشمندانه روشهای مختلف مدلسازی و استفاده از اطلاعات متنوع برای تولید مدلهای با کیفیت بالاست. علیرغم ظهور ابزارهای قدرتمندی مانند AlphaFold 2، I-TASSER همچنان به دلیل جامعیت، انعطافپذیری و قابلیتهای آنالیز عملکردی، یکی از انتخابهای اصلی محققان در سراسر دنیاست. با پیشرفتهای اخیر در ترکیب آیتاسر با روشهای یادگیری عمیق، انتظار میرود این ابزار همچنان در خط مقدم تحقیقات بیوانفورماتیک باقی بماند.
برای محققان و دانشجویان علاقهمند به بیوانفورماتیک و طراحی دارو، تسلط بر استفاده از آیتاسر و تفسیر صحیح نتایج آن، یک مهارت ارزشمند محسوب میشود. با توجه به اهمیت روزافزون ساختار پروتئین در درک مکانیسمهای مولکولی بیماریها و توسعه داروهای جدید، آیتاسر به عنوان یک ابزار کارآمد و قابل دسترس، نقش کلیدی در پیشبرد این حوزههای تحقیقاتی خواهد داشت. با ادغام بیشتر فناوریهای نوین مانند یادگیری عمیق و استفاده از دادههای بیولوژیکی متنوع، نسلهای آینده I-TASSER میتوانند به ما در حل چالشهای پیچیدهتر بیولوژیکی کمک کنند.
I-TASSER یکی از پیشرفتهترین ابزارهای پیشبینی ساختار سهبعدی پروتئین است. این ابزار از توالی اسیدهای آمینه پروتئین برای شناسایی الگوهای ساختاری موجود، مونتاژ ساختارهای سهبعدی، پالایش مدلها، و پیشبینی عملکرد پروتئین استفاده میکند. کاربرد اصلی آن در درک عملکرد پروتئینها، طراحی دارو، و مطالعات بیوانفورماتیک ساختاری است.
I-TASSER به عنوان یکی از دقیقترین ابزارهای پیشبینی ساختار پروتئین شناخته میشود. این ابزار در رقابتهای جهانی CASP، که برای ارزیابی روشهای پیشبینی ساختار پروتئین برگزار میشود، عملکرد بسیار بالایی داشته است. دقت پیشبینی به کیفیت الگوهای ساختاری موجود در پایگاه داده PDB و پیچیدگی پروتئین بستگی دارد. برای پروتئینهایی که الگوهای مناسبی دارند، دقت بسیار بالا است.
برای استفاده از آیتاسر، کاربران باید توالی پروتئین خود را در قالب FASTA به سرور آنلاین آیتاسر ارسال کنند. این ابزار به صورت خودکار فرآیند پیشبینی را آغاز کرده و نتایج شامل مدلهای سهبعدی، نمرات اعتبارسنجی، و پیشبینی عملکرد پروتئین را ارائه میدهد. برای دسترسی به سرور، میتوانید به وبسایت رسمی آیتاسر مراجعه کنید.
برای ثبتنام و استفاده از آیتاسر شما نیازمند یک ایمیل سازمانی میباشید، ایمیلهایی با پسوند gmail ،outlook و … قابل قبول نمیباشند.
بله، سرور آنلاین I-TASSER به صورت رایگان برای استفاده عمومی در دسترس است. با این حال، کاربران میتوانند نسخههای مستقل نرمافزار را برای اجرا بر روی سیستمهای محاسباتی شخصی دریافت کنند، که ممکن است نیاز به تنظیمات پیشرفته داشته باشد.

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
سلام، وقت بخیر.
چطور میتونیم بهتون کمک کنیم؟ 🤓
تیم ما آماده پاسخگویی به سوالات شماست.
پشتیبانی 24 ساعته در 7 روز هفته.