دوره NGS منتشر شد …

زمان تخمینی مطالعه: 14 دقیقه
NCBI (مرکز ملی اطلاعات بیوتکنولوژی) یک سازمان تحقیقاتی مستقر در ایالات متحده است که دسترسی به پایگاههای داده و ابزارهای مختلف برای اطلاعات زیستپزشکی و ژنومی را فراهم میکند. یکی از ابزارهای مبتنی بر وب و کاربردی که NCBI ارائه میدهد، Primer BLAST است که میتواند به شما در طراحی و ارزیابی پرایمرهای PCR کمک کند.
Primer-BLAST از دو الگوریتم استفاده میکند: Primer3 و BLAST
Primer3، نرمافزاری است که پرایمرهای PCR را بر اساس معیارهای مختلفی مانند طول پرایمر، دمای ذوب، محتوای GC و تشکیل ساختار ثانویه، طراحی و آنالیز میکند.
BLAST، نرمافزاری است که توالیهای مشابه را در یک پایگاهداده یا دیتابیس جستجو میکند و میزان اختصاصیت (specificity) پرایمرها را بررسی میکند.
* Primer-BLAST به شما این امکان را میدهد تا پرایمرهای اختصاصی برای توالی هدف خود را در یک مرحله طراحی کنید یا اختصاصیت پرایمرهایی که طراحی کردهاید را بررسی کنید.
** مشابه سایر جستجوهای BLAST، میتوانید جستجوی Primer-BLAST را به گونههای خاص یا نواحی خاصی محدود کنید.
*** Primer-BLAST بهعنوان خروجی، پرایمرهای کاندید را همراه با همترازی (alignment) آنها با الگو (template) ارائه میدهد.
در این بخش میخواهیم شما را با قسمتهای مختلف صفحه Primer BLAST در سایت NCBI آشنا کنیم.
صفحه Primer BLAST تنظیمات طراحی پرایمر را به دو قسمت کلی تقسیم میکند: تنظیمات عمومی و تنظیمات پیشرفته
برای ورود به نرمافزار Primer BLAST میتوانید از لینک زیر استفاده نمایید.
با ورود به Primer BLAST صفحهای را مشاهده میکنید که بایستی تنظیمات آن را در طراحی پرایمرهای اختصاصی خود تغییر دهید.
پیشنهاد: دوره جامع طراحی پرایمر یک فرصت طلایی برای یادگیری حرفهای طراحی پرایمر با نرمافزار Primer BLAST و سایر ابزارهای پرکاربرد در این حوزه است.

این بخش در بالاترین قسمت از صفحه Primer BLAST قرار میگیرد:

حالت A برای طراحی پرایمر در زمانی است که یک الگو برای تکثیر دارید و هدف شما هم همان الگو است.
انتخاب حالت B برای زمانی است که میخواهید جفت پرایمرهایی برای هدفگیری چند توالی طراحی کنید.
در بخش C میتوانید توالی ناحیه ژنومیک مورد نظر خود (که میخواهید برای آن پرایمر طراحی کنید) را به فرمت FASTA وارد کنید. همچنین میتوانید کد دسترسی (ID) آن ناحیه را وارد کنید یا فایل FASTA توالی خود را از قسمت Browse آپلود کنید.
دربخش D میتوانید تعیین کنید که نرمافزار از کجا تا کجای توالی شما را برای طراحی پرایمر فوروارد (Forward) یا ریورس (Reverse) در نظر بگیرد.
پیشنهاد: برای آشنایی با طراحی پرایمر مقاله، طراحی پرایمر چیست؟ طراحی پرایمر به زبان ساده و کاربردی برای PCR مطالعه کنید.
در این قسمت، میتوانید پارامترهای مربوط به پرایمرها را تعیین کنید:

در بخش E میتوانید توالی پرایمرهای موردنظر خود را برای چککردن اختصاصیت وارد کنید. جهت پرایمرها را باید از ‘5 به ‘3 بنویسید.
در قسمتهای بعدی بهترتیب: تعیین سایز محصول PCR، تعداد پرایمرهایی که نرمافزار در هر اجرا به شما برمیگرداند، و دمای ذوب پرایمر (Tm) را میتوانید تعیین کنید. البته برای همه این موارد، مقادیر default یا مشخصشده مناسب هستند.
پینشهاد: اگر قصدداری توالی مورد نظرت رو قبل از وارد کردن به طراحی پرایمر، ویرایش کنی و بخش های اضافیش رو حذف کنی میتونی از Vanyar Nucleotide Sequence Editor استفاده کنی.
این بخش مربوط به زمانی است که مولکول RNA را بهعنوان یک الگو برای PCR انتخاب میکنید، یعنی میخواهید از RT-PCR یا Reverse Transcription استفاده کنید:

مشکل ما در عمده RT-PCRها این است که وقتی RNA استخراج میکنیم، معمولا مقداری DNA ژنومی هم با آن استخراج میشود. حالا اگر پرایمرها علاوه بر cDNA به DNA ژنومی هم وصل شوند، درواقع آلودگی DNA هم داریم ولی ما متوجه آن نمیشویم.
Primer BLAST در بخش Exon/intron selection میتواند اتصالات اگزونی را (Exon junction) برای طراحی پرایمر در نظر بگیرد. در قسمت (F)، Exon junction span، میتوانید طوری تنظیم کنید که پرایمرهای کاندید روی محل اتصال اگزونها قرار بگیرند یا نگیرند.
همچنین میتوانید با فعالکردن قسمت (G) Intron inclusion، تایید کنید که جفت پرایمرها باید به اندازه حداقل یک اینترون بر روی توالی ژنومیک از هم فاصله داشته باشند. این آپشن اجازه میدهد تا با طویل شدن توالی بهخاطر اینترون، PCR مطلوبی روی DNA ژنومی (آلودگی ژنومی) رخ ندهد؛ چراکه سایز محصول خیلی بالا خواهد بود و فقط cDNA تکثیر میشود.
پیشنهاد: بهمنظور ثبت سفارش طراحی پرایمر به صفحه تهیه پلنهای طراحی پرایمر و پروب مراجعه فرمایید. طراحی پرایمر در میزکار وانیار و زیرنظر کارشناسان بیوانفورماتیک صورت میگیرد.
در این قسمت با پارامترهای بررسی اختصاصیت برای جفتپرایمرها مواجه میشویم:
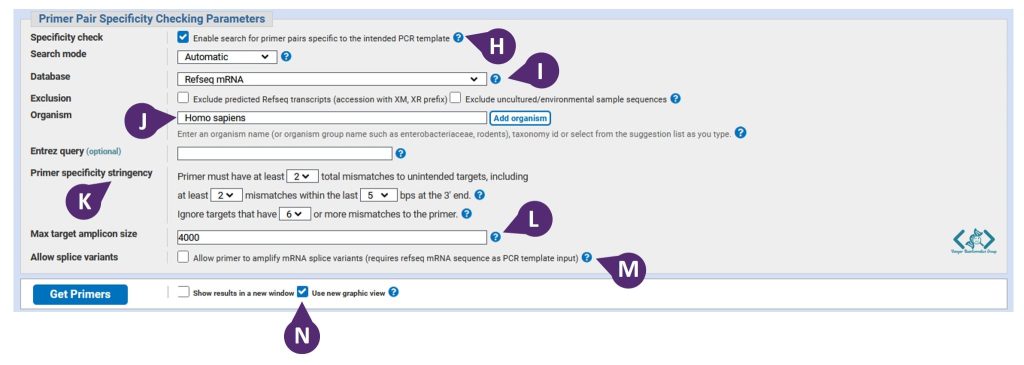
بخش H: پرایمرهایی specific یا اختصاصی براساس دیتابیسی که به نرم افزار بدهید، انتخاب میشوند.
بخش I: انتخاب دیتابیس، که اگر بر روی DNA ژنومیک کار میکنید، برای موجوداتی که ژنوم رفرنس دارند از دیتابیسهای Refseq representative genomes و Genomes for selected organisms استفاده میکنیم، و برای موجوداتی که ژنوم رفرنس ندارند از nr استفاده میکنیم. (nr هم دارای RNA هست و هم DNA، هم رفرنس و هم غیر رفرنس). اگر با cDNA کار میکنید، بایستی از دیتابیس Refseq mRNA یا Refseq RNA استفاده کنید.
بخش J: از قسمت Organism میتوانید جستجو را به موجود خاصی محدود کنید.
بخش K: تعیین یک سری پارامتر برای اعمال سختگیری در اختصاصیت پرایمرها.
اما این اعداد به چه معنا هستند؟
*** پرایمرهایی که mismatch یا عدم تطابق آنها با اهداف ناخواسته (unintended targets) در حداقل 2 تا نوکلئوتید باشد و این 2 تا در 5 تا نوکلئوتید آخر انتهای ‘3 باشند را میتوانیم بپذیریم. هرچه mismatch پرایمر با اهداف ناخواسته بیشتر باشد، پرایمر اختصاصیتر است. پس هرچقدر این مقادیر را بالاتر ببریم، سختگیرانهتر است.
*** اگر پرایمر با اهداف ناخواسته ۶ تا یا بیشتر نوکلئوتید عدم تطابق داشت، آن هدف را دیگر درنظر نمیگیریم، یعنی پرایمر آن را نمیشناسد. هرچه این عدد پایینتر باشد، پرایمرها با mismatchهای کمتری انتخاب میشوند (سختگیری بیشتر).
بخش L: قسمت Max target amplicon size نشان میدهد که محصولات PCR بزرگتر از 4000bp هیچ مزاحمتی برای تست شما ایجاد نمیکنند. این مورد قابل تغییر است.
بخش M: اگر گزینه Allow splice variants را تیک بزنید، اجازه طراحی جفت پرایمرهایی را میدهید که همه گونههای رونوشت شناختهشده (transcript variants) را برای یک ژن تکثیر میکنند.
بخش N: نتایج طراحی پرایمر را میتوانید با انتخاب این گزینه بهصورت تصویری مشاهده کنید.
پیشنهاد: افزایش فرصتهای شغلی و تحقیقاتی با شرکت در دوره بیوانفورماتیک عمومی و کسب مهارتهای اساسی بیوانفورماتیک.

با کلیک بر روی گزینه “Advanced parameters” بخشی را باز میکنید که تنظیمات آن را در طراحی پرایمر بهندرت تغییر میدهیم:
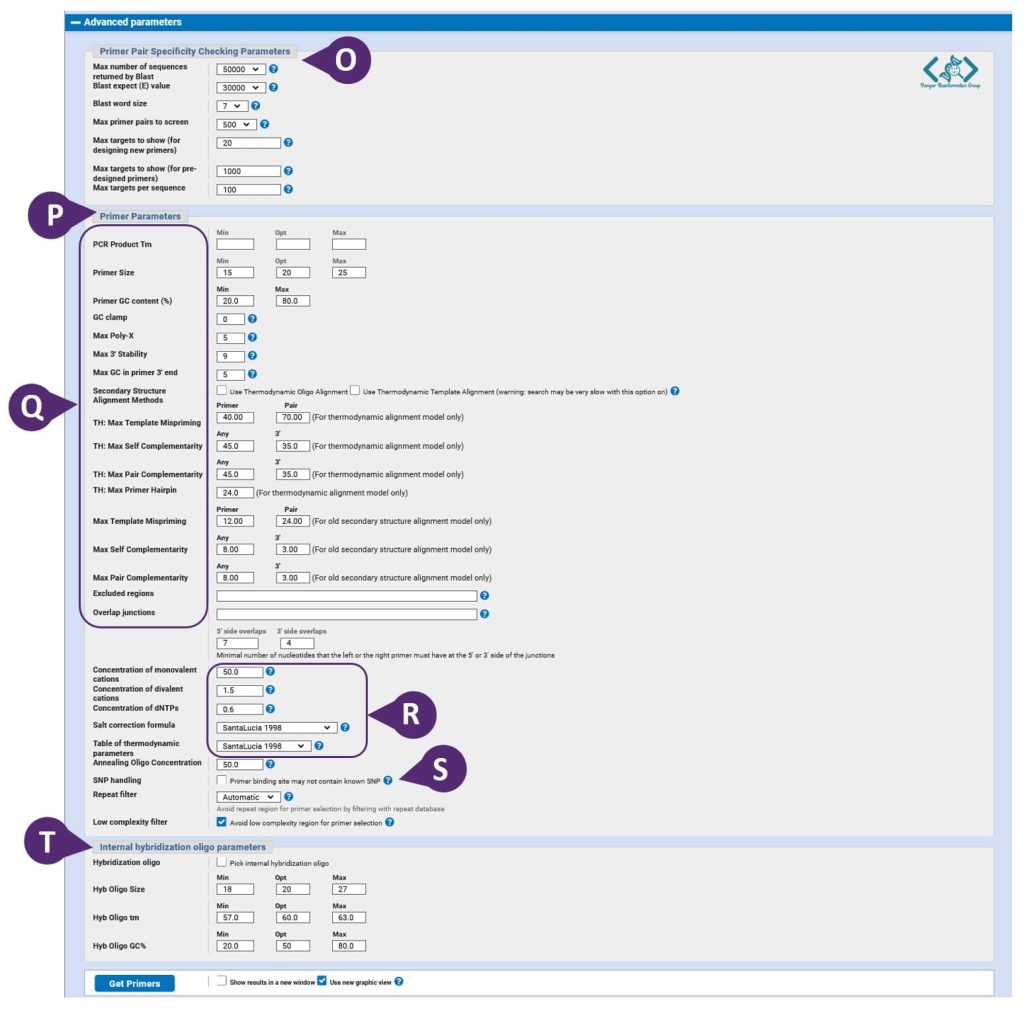
بخش اول (O) شامل پارامترهایی برای BLAST است که اختصاصیت پرایمرها را بررسی میکند.
بخش دوم (P) شامل پارامترهای مخصوص پرایمرهای انتخابشده و محصولات PCR آنها است: مانند Tm محصول PCR، طول پرایمر، محتوای GC پرایمر و گیرههای GC یا GC clamp در انتهای ‘3 پرایمر (قسمت Q). همچنین شامل تنظیماتی برای شرایط بافر PCR است (قسمت R) زیرا این شرایط میتواند تا حد زیادی بر محاسبه Tm پرایمر تأثیر بگذارد.
با علامت زدن کادر (S) به Primer-BLAST دستور میدهید تا SNPهای مربوط به توالی DNA الگو را در هنگام انتخاب پرایمر در نظر بگیرد و در نواحی با SNPهای با فراوانی جمعیتی بالا پرایمر طراحی نکند.
با فعال کردن و تنظیم گزینههای دادهشده در بخش سوم (T) میتوانید پروب داخلی برای Real-time PCR انتخاب کنید.
در این قسمت مقاله، پلهبهپله مراحل طراحی پرایمر را با یک مثال آموزش میدهیم:
در این مثال، فرض کنید که میخواهیم کل اگزون ۳ از ژن SLC25A5 انسانی را تکثیر کنیم (یعنی روی DNA کار میکنیم نه mRNA). پس بایستی برای دو طرف این اگزون پرایمر طراحی کنیم. اما چگونه؟
ابتدا مانند تصویر زیر، اسم ژن SLC25A5 را در NCBI و بخش Gene وارد میکنیم و سپس بر روی Search کلیک میکنیم:
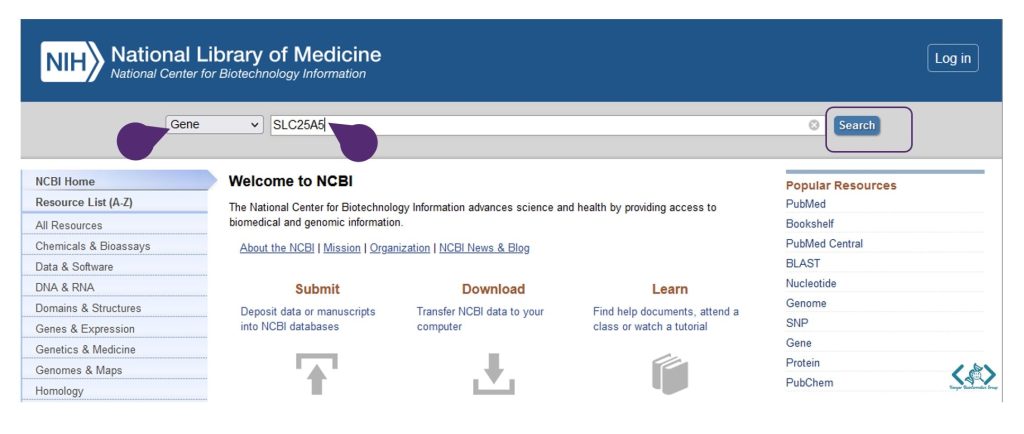
حالا بر روی ژن SLC25A5 انسانی (Homo sapiens) کلیک میکنیم:
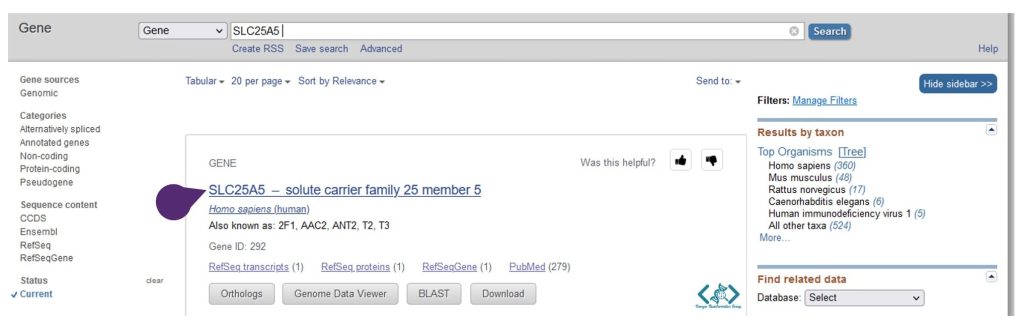
سپس از قسمت NCBI Reference Sequences (RefSeq)، و بخش Genomic، روی گزینه Sequence Viewer (Graphics) کلیک میکنیم تا نمای گرافیکی ژن موردنظر خود را ببینیم:
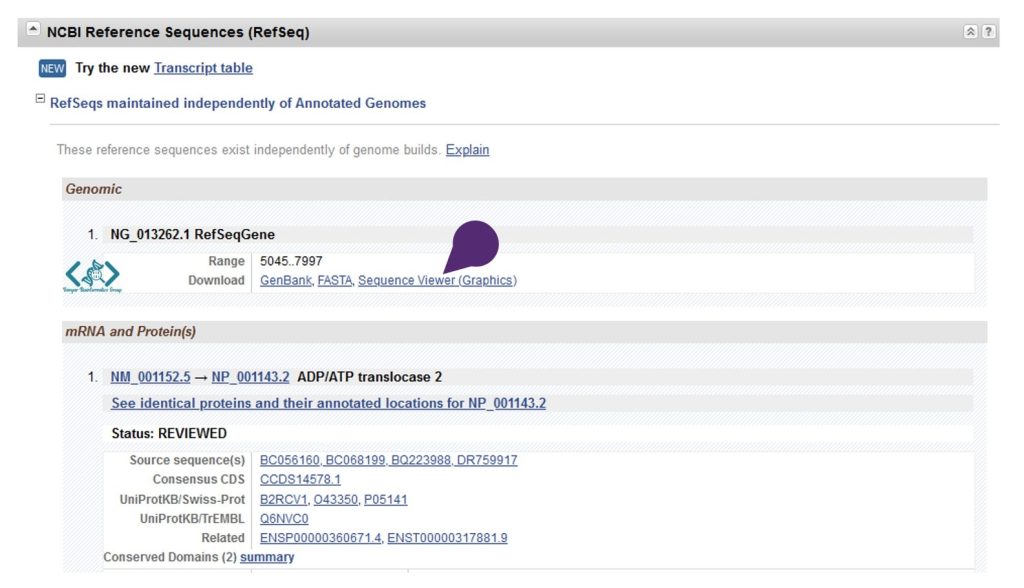
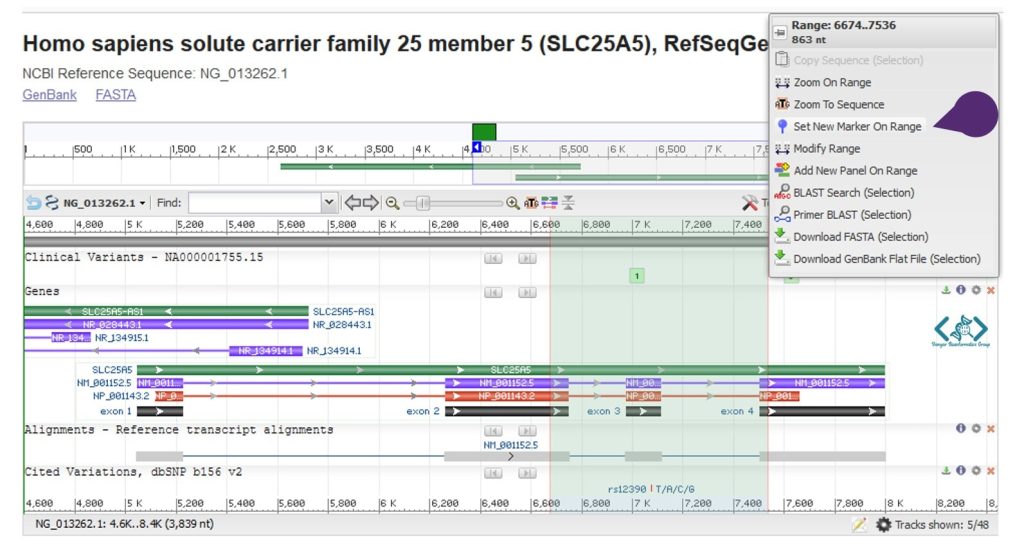
بعد ناحیه اطراف اگزون ۳ را با گرفتن کلیکچپ موس انتخاب میکنیم و با رها کردن آن، بر گزینه Set New Marker On Range میزنیم تا این ناحیه به عنوان مارکر درنظر گرفته شود.
دقت کنید که طول قطعهای که در اینجا انتخاب کردیم، ۸۶۳ نوکلئوتید است:
حالا میتوانید از بخش دانلود، توالی FASTA این مارکر را دانلود کنید:
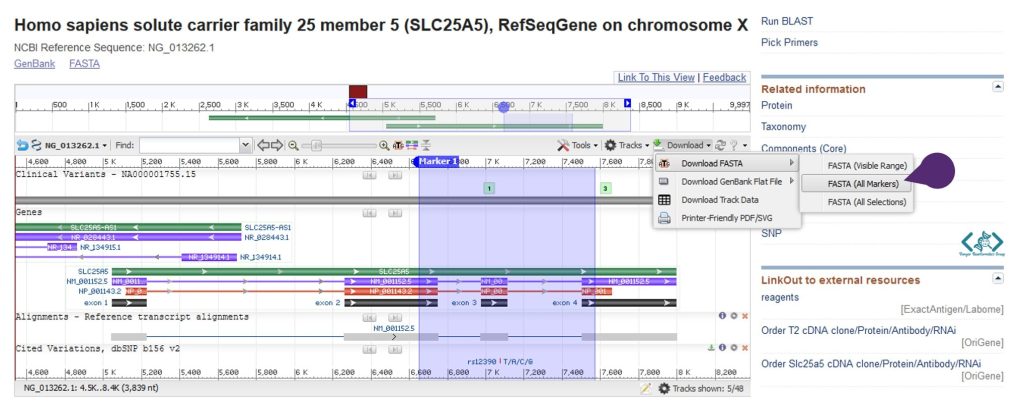
اگزون موردنظر ما ۱۴۱ نوکلئوتید طول دارد. این را با قراردادن موس روی اگزون ۳ متوجه میشوید.
برای تکثیر اگزون و دریافت توالی کامل آن طی سکانس، باید پرایمرها با حدود ۳۰ نوکئوتید فاصله نسبت به ابتدا و انتهای اگزون طراحی شوند.
با این اوصاف میتوانیم محدوده طراحی پرایمرهای فوروارد و ریورس را تعیین کنیم:
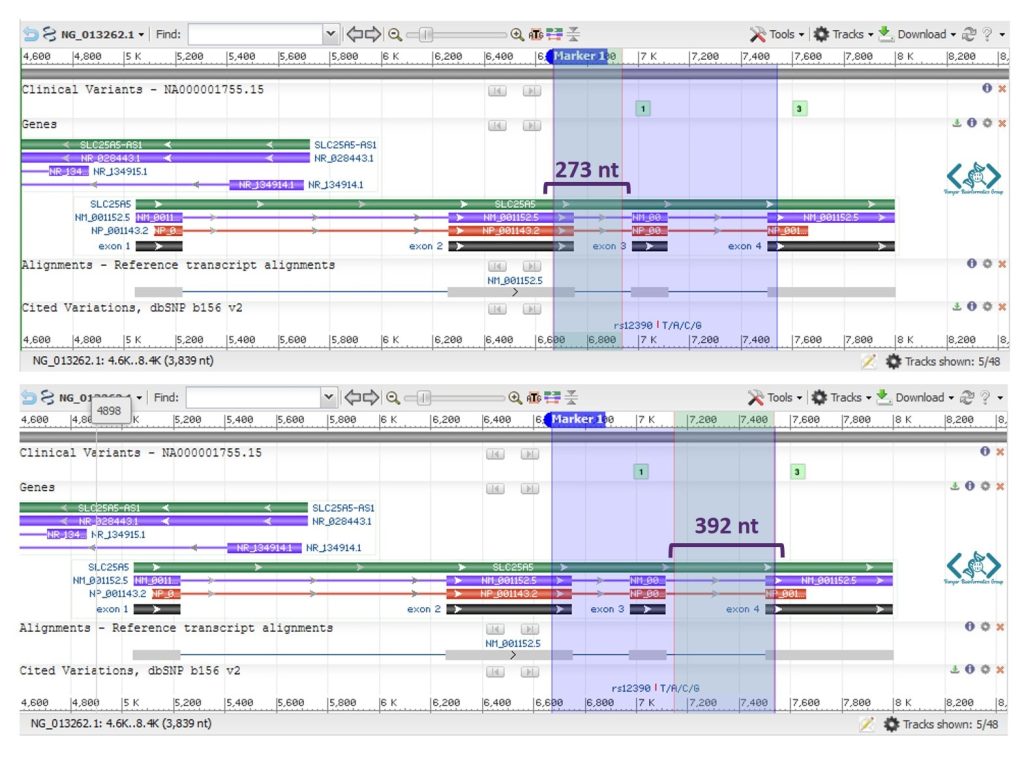
فاصلهای که اینجا در نظر گرفته ایم: ۲۷۳ تا نوکلئوتید فاصله از سمت چپ و ۳۹۲ تا نوکلئوتید تا آخر توالی است.
حالا باید به صفحه Primer BLAST بروید و توالی خود را در اختیار نرمافزار قرار دهید تا پرایمرهای اختصاصی را برای شما طراحی کند.
میتوانید توالی فایل FASTA را در Notepad سیستم خود ببینید و از آنجا copy کرده و در باکس نرمافزار paste کنید. در این صفحه باید محدوده طراحی پرایمر را هم تعیین کنید و سایر پارامترهای موردنظر خود را تغییر دهید:
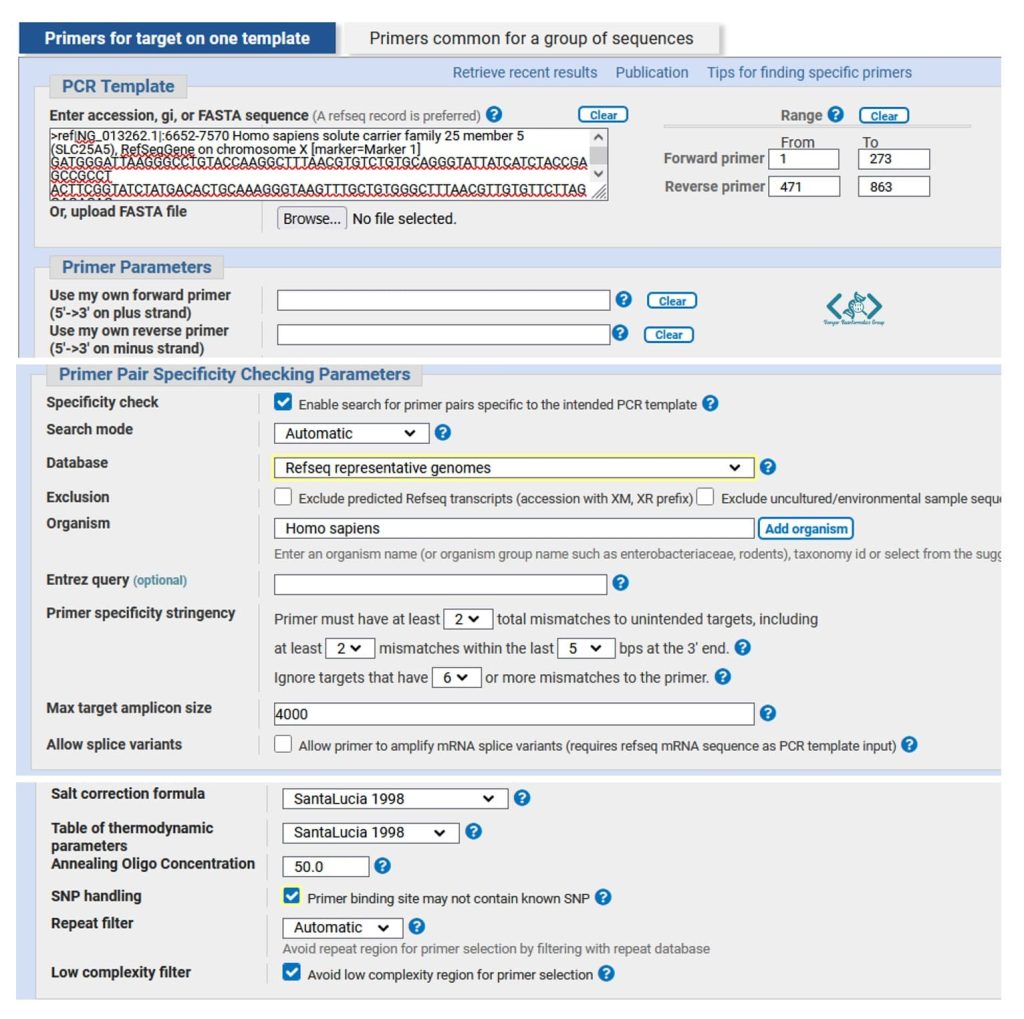
بعد از کلیک بر روی Get Primers در انتهای صفحه، با چنین پنجرهای روبهرو میشوید:
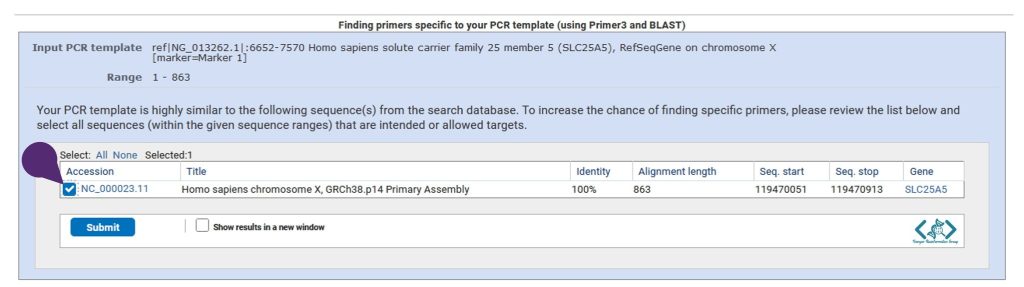
در این صفحه تایید میکنید که میخواهید برای ژنی که نرمافزار تشخیص داده، طراحی پرایمر کنید.
پیشنهاد: مقاله طراحی پرایمر با نرمافزار Oligo 7 را مطالعه کنید.
حالا بعد از تایید، باید کمی صبر کنید تا Primer BLAST بهترین پرایمرها را برای ناحیه انتخابی شما طراحی کند و چنین خروجی را به شما نمایش دهد:
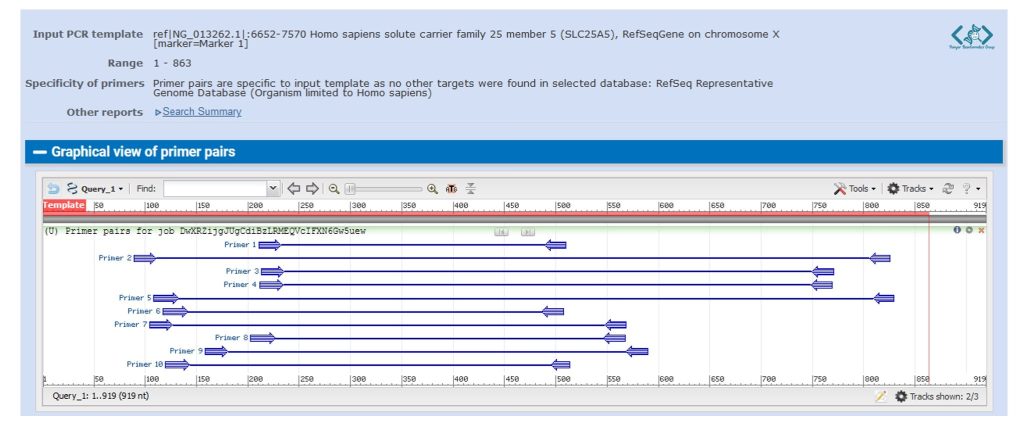
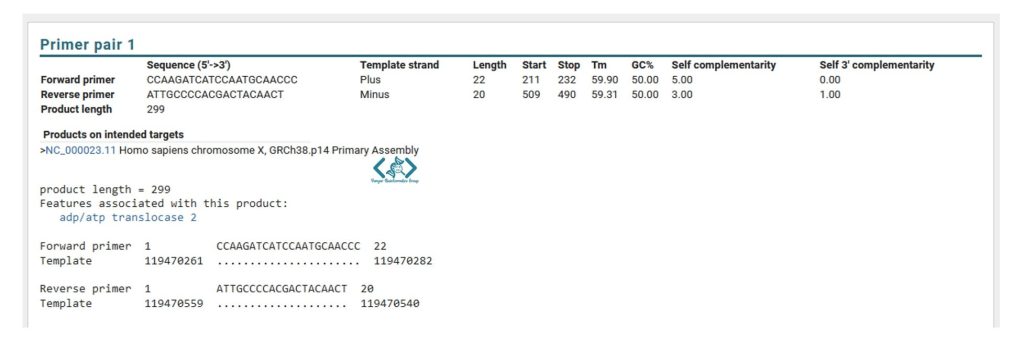
با اسکرول کردن به پایین صفحه، خصوصیات و توالی همه جفت پرایمرهای اختصاصی برای توالی خود را میتوانید مشاهده کنید و از آنها استفاده نمایید:
در این مقاله از مجله بیوانفورماتیک وانیار، به معرفی ابزار Primer BLAST پرداختیم و پارامترهای قابل تنظیم آن را شرح دادیم. بهعلاوه، نحوه استفاده از Primer BLAST را با یک مثال بهشکل عملی و گامبهگام توضیح دادیم.
Primer BLAST ابزاری قدرتمند است که حتما در طراحی پرایمر PCR به آن احتیاج پیدا میکنید؛ و ما بسیار خوشحال میشویم اگر تجربه و چالشهای خود را حین استفاده از این ابزار با وانیار به اشتراک بگذارید :)))
Primer BLAST ابزاری تحتوب در سایت NCBI است که از دو الگوریتم Primer3 و BLAST استفاده میکند تا به شما در طراحی و ارزیابی پرایمرهای PCR کمک کند.
بله Primer BLAST یک ابزار کاملا رایگان و آنلاین در مرکز بیوتکنولوژی آمریکا است که برای عموم از سایت NCBI در دسترس میباشد.
بله با استفاده از ابزار پرایمر بلاست (Primer BLAST) شما می توانید برای روشهای مختلف PCR پرایمرهایی با درجه اختصاصیت بالا و کارایی مناسب تولید نمایید.

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
سلام، وقت بخیر.
چطور میتونیم بهتون کمک کنیم؟ 🤓
تیم ما آماده پاسخگویی به سوالات شماست.
پشتیبانی 24 ساعته در 7 روز هفته.