دوره NGS منتشر شد …

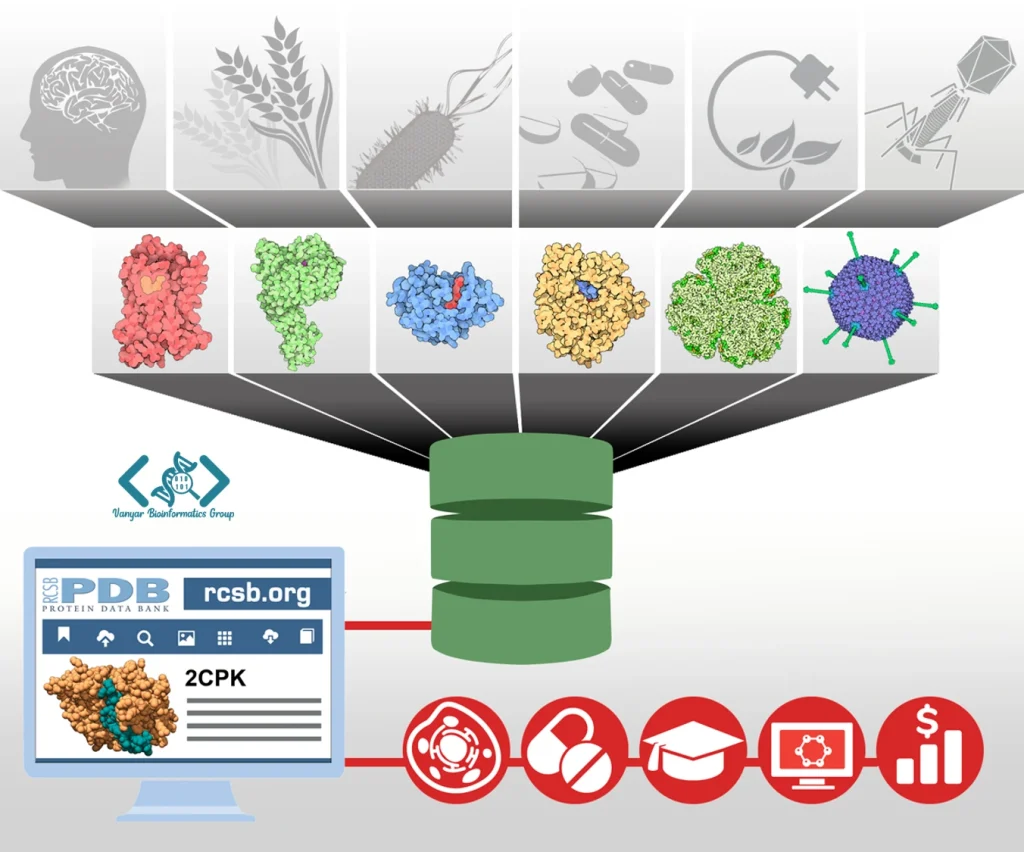
PDB یا Protein Data Bank (بانک اطلاعاتی پروتئین) یکی از ارکان اساسی در حوزه بیوانفورماتیک ساختاری است که دارای دو مفهوم کلیدی میباشد. از یک سو، PDB به یک پایگاه داده جهانی برای ذخیره اطلاعات ساختار سهبعدی ماکرومولکولهای زیستی اشاره دارد و از سوی دیگر، به فرمت استانداردی برای ذخیره این دادههای ساختاری اطلاق میشود. درک صحیح این دو مفهوم برای متخصصان بیوانفورماتیک، زیستشناسان ساختاری و محققان طراحی دارو ضروری است. در این مقاله، به بررسی جامع هر دو جنبه PDB میپردازیم و کاربردهای متنوع آن را در تحقیقات علمی و صنعت داروسازی شرح میدهیم.
پایگاه داده PDB در سال 1971 با تنها 7 ساختار پروتئینی آغاز به کار کرد. این ابتکار علمی توسط گروهی از محققان در آزمایشگاه بروکهیون بنیانگذاری شد و هدف اصلی آن ذخیره و به اشتراکگذاری دادههای کریستالوگرافی اشعه ایکس بود. در طول دهههای بعدی، با پیشرفت تکنیکهای تعیین ساختار مانند NMR و اخیراً میکروسکوپ کرایوالکترونی (Cryo-EM)، این پایگاه داده به طور چشمگیری گسترش یافت. امروزه، مدیریت بانک اطلاعاتی پروتئین توسط کنسرسیوم بینالمللی wwPDB صورت میگیرد که شامل مراکز اصلی در آمریکا (RCSB PDB)، اروپا (PDBe)، ژاپن (PDBj) و مرکز اطلاعات زیستمولکولی چین (BMCD) است.
فرمت فایل PDB همزمان با رشد دیتابیس توسعه یافت. این فرمت در ابتدا برای ذخیره مختصات اتمی پروتئینهای کریستالوگرافیشده طراحی شد و با گذر زمان برای پوشش انواع ماکرومولکولها و روشهای تجربی گسترش یافت. محدودیتهای فرمت اصلی PDB، مانند محدودیت در تعداد اتمها و نمایش سلولهای واحد، منجر به توسعه فرمتهای تکمیلی مانند mmCIF شد. با این حال، فرمت کلاسیک PDB به دلیل سادگی و قابلیت خوانش توسط انسان، همچنان استاندارد عملی در بسیاری از نرمافزارهای بیوانفورماتیک باقی مانده است.
بانک اطلاعاتی پروتئین یک مخزن سازمانیافته از ساختارهای ماکرومولکولی است. هر ساختار در این پایگاه با یک کد شناسایی منحصربهفرد چهار کاراکتری (مانند 4HHB برای هموگلوبین انسانی) مشخص میشود. دادههای موجود در بانک اطلاعاتی پروتئین از طریق واسطهای کاربری وب مانند RCSB PDB، PDBe و PDBj قابل جستجو و دسترسی هستند. این پایگاه امکان جستجو بر اساس پارامترهای متعدد مانند نام پروتئین، عملکرد بیولوژیکی، ارگانیسم منبع، روش تعیین ساختار و خصوصیات ساختاری را فراهم میکند. کاربران میتوانند ساختارها را در فرمتهای مختلف دانلود کرده و با استفاده از ابزارهای تجسم آنلاین مشاهده نمایند.
دیتابیس PDB صرفاً محدود به پروتئینها نیست و مجموعه متنوعی از ماکرومولکولهای زیستی را در بر میگیرد. این پایگاه شامل ساختارهای اسیدهای نوکلئیک (DNA و RNA)، کمپلکسهای پروتئین-اسید نوکلئیک، پروتئین-لیگاند و اخیراً حتی ویروسهای کامل میباشد. هر رکورد بانک اطلاعاتی پروتئین حاوی اطلاعات متنوعی است که فراتر از مختصات اتمی میباشد: دادههای آزمایشگاهی استفادهشده برای تعیین ساختار، شرایط آزمایش، اطلاعات توالی، اطلاعات مرجع، عملکرد بیولوژیکی و ارتباطات با سایر پایگاههای داده زیستی. این طیف وسیع از دادهها، بانک اطلاعاتی پروتئین را به یک منبع ارزشمند برای تحقیقات میانرشتهای تبدیل کرده است.
کیفیت دادههای موجود در بانک اطلاعاتی پروتئین از اهمیت بالایی برخوردار است، زیرا مطالعات ساختاری و طراحی دارو بر پایه این اطلاعات انجام میشوند. برای اطمینان از کیفیت دادهها، wwPDB فرآیند اعتبارسنجی جامعی را برای ساختارهای جدید اجرا میکند. این ارزیابی شامل بررسی هندسه شیمیایی، تطابق با دادههای تجربی، مقایسه با ساختارهای مشابه و ارزیابی نقشههای الکترون دانسیته است. مقادیر شاخصهای کیفیت مانند R-factor، Free R-factor و Ramachandran plot برای هر ساختار گزارش میشود. کاربران میتوانند با استفاده از این معیارها، ساختارهای با کیفیت بالاتر را برای مطالعات خود انتخاب کنند.
فایل PDB یک فایل متنی با فرمت ثابت است که اطلاعات ساختاری ماکرومولکولها را ذخیره میکند. این فایل از خطوطی تشکیل شده که هر کدام با یک کلیدواژه یا “رکورد” شروع میشوند. رکوردهای اصلی شامل HEADER (اطلاعات شناسایی)، TITLE (عنوان ساختار)، AUTHOR (نویسندگان)، REMARK (اطلاعات تکمیلی)، SEQRES (توالی آمینواسیدی)، ATOM (مختصات اتمهای اصلی) و HETATM (مختصات مولکولهای غیر پروتئینی مانند لیگاندها) میباشند. هر خط دارای ساختار دقیقی است که در آن هر ستون معنای خاصی دارد. این ساختار استاندارد باعث میشود فایلهای PDB توسط طیف گستردهای از نرمافزارها قابل تفسیر باشند.
رکوردهای ATOM و HETATM اصلیترین بخشهای یک فایل PDB هستند که مختصات سهبعدی ساختار را مشخص میکنند. هر خط ATOM حاوی اطلاعاتی مانند شماره اتم، نام اتم، نام آمینواسید، زنجیره، شماره آمینواسید در زنجیره، و مختصات X، Y، Z است. فاکتور B یا فاکتور دمایی نیز در این رکوردها ذخیره میشود که نشاندهنده میزان تحرک اتم است. رکوردهای CONECT اطلاعات پیوند شیمیایی بین اتمها را مشخص میکنند که به خصوص برای لیگاندها و مولکولهای کوچک مهم است. بخش SEQRES توالی اصلی پروتئین را نشان میدهد که میتواند با ساختار موجود در مختصات اتمی متفاوت باشد، زیرا برخی قسمتهای پروتئین ممکن است در ساختار کریستالی قابل مشاهده نباشند.
در این جا قصد داریم ساختار پروتئین را با کددسترسی PDB جستجو و فایل ساختار سه بعدی آن را در قالب PDB از پایگاه دریافت نماییم، ابتدا از طریق لینک زیر به پایگاه PDB وارد میشویم:
در صفحه ابتدای سایت با کادر جستجو در پایگاه روبهرو خواهیم شد، در این کادر میتوانیم توالی، نام مولکول یا ID دسترسی به مولکول مورد نظر را وارد نماییم.

Include CSM با فعال کردن این گزینه نتایج جستجو علاوه بر ساختارهای تجربی شامل ساختارهای محاسباتی هم خواهد بود.
در این مثال قصد داریم پروتئینی با کد دسترسی 8ZR2 را جستجو کنیم.
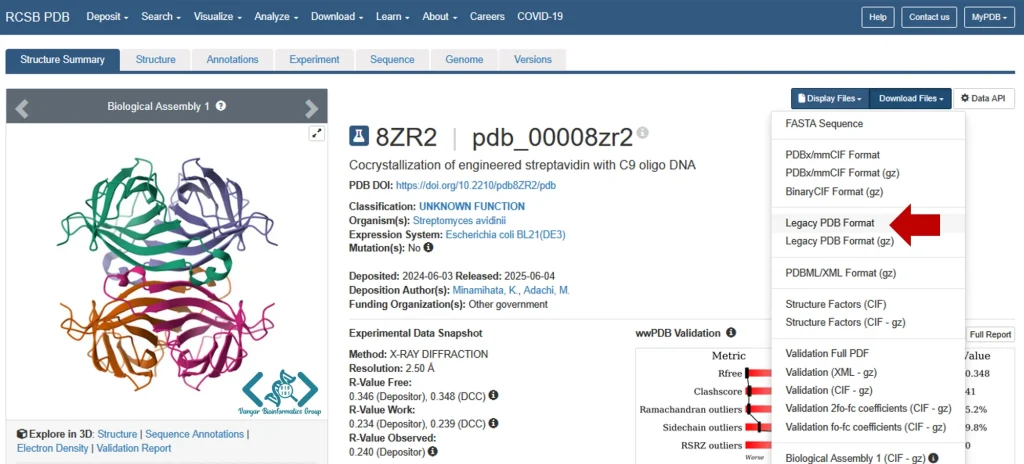
Download File از طریق این دکمه میتوان فایل فرمتهای مختلف ساختاری از جمله فایل PDB را برای مولکول مورد نظر دانلود نمود.
با وجود کاربرد گسترده، فرمت PDB دارای محدودیتهایی است. یکی از مهمترین محدودیتها، ناتوانی در ذخیره ساختارهای بزرگ با بیش از 99,999 اتم است. این مشکل در مطالعه ساختارهای پیچیدهای مانند ریبوزومها یا ویروسهای کامل چالشبرانگیز است. محدودیت دیگر، نمایش پیوندهای شیمیایی است که در فرمت PDB به صورت کامل قابل نمایش نیست. همچنین، این فرمت برای ذخیرهسازی برخی اطلاعات آزمایشگاهی خاص مناسب نیست. به دلیل این محدودیتها، فرمتهای جایگزین مانند mmCIF توسعه یافتهاند که انعطافپذیری بیشتری دارند. با این وجود، به دلیل سادگی و قابلیت خوانش توسط انسان، فرمت PDB همچنان به طور گسترده استفاده میشود.
نرمافزارهای تجسم مولکولی ابزارهای کلیدی برای مشاهده و تفسیر ساختارهای PDB هستند. PyMOL یکی از محبوبترین این نرمافزارهاست که امکان نمایش با کیفیت بالا، تولید تصاویر و انیمیشن، و تحلیل ساختاری را فراهم میکند. UCSF Chimera با قابلیتهای پیشرفته در زمینه محاسبات ساختاری و ادغام دادههای چندگانه شناخته میشود. VMD (Visual Molecular Dynamics) برای شبیهسازی دینامیک مولکولی و تجسم ساختارهای بزرگ بهینه شده است. Swiss-PDB Viewer (Deep View) نرمافزاری سبک و کاربرپسند است که برای مدلسازی همولوژی و تحلیل ساختاری مناسب است. Jmol/JSmol به عنوان یک نمایشگر مبتنی بر Java/JavaScript امکان تجسم ساختارهای PDB را در مرورگرهای وب فراهم میکند.
برای تحلیل و دستکاری فایلهای PDB، ابزارهای تخصصی متعددی وجود دارند. Biopython در زبان Python و Bio3D در R، کتابخانههای قدرتمندی برای پردازش خودکار فایلهای PDB هستند. DSSP برای محاسبه ساختارهای ثانویه از مختصات اتمی استفاده میشود. PDBSUM خلاصههای گرافیکی جامعی از ساختار و عملکرد پروتئینها ارائه میدهد. PDB-tools مجموعهای از اسکریپتهای خط فرمان برای دستکاری سریع فایلهای PDB است. ProCheck و WHAT_CHECK برای بررسی هندسه و کیفیت ساختارهای پروتئینی کاربرد دارند. MolProbity تحلیل جامعی از تداخلهای اتمی و زوایای پیوندی ارائه میدهد که برای ارزیابی کیفیت ساختاری بسیار مفید است.
کتابخانههای برنامهنویسی نقش مهمی در تحلیل خودکار و پردازش حجم بالای دادههای بانک اطلاعاتی پروتئین دارند. کتابخانه Bio.PDB در Biopython امکان خواندن، نوشتن و دستکاری فایلهای PDB را با انعطافپذیری بالا فراهم میکند. ProDy یک کتابخانه Python است که برای تحلیل دینامیک پروتئین و مدلسازی نرمال مود بهینه شده است. BioPandas ادغام دادههای ساختاری PDB با قابلیتهای تحلیل داده pandas را امکانپذیر میسازد. MDAnalysis برای تحلیل شبیهسازیهای دینامیک مولکولی طراحی شده و میتواند با فایلهای PDB کار کند. کتابخانه OpenStructure یک چارچوب محاسباتی برای زیستشناسی ساختاری با تمرکز بر پروتئینها و تعاملات آنها ارائه میدهد.
ساختارهای PDB نقش حیاتی در مدلسازی همولوژی و پیشبینی ساختار پروتئین ایفا میکنند. در مدلسازی همولوژی، ساختارهای موجود در PDB به عنوان الگو برای پیشبینی ساختار سهبعدی پروتئینهای با توالی مشابه استفاده میشوند. نرمافزارهایی مانند MODELLER و SWISS-MODEL به طور گسترده از این رویکرد استفاده میکنند. در روشهای threading یا تطبیق تاخوردگی، ساختار یک پروتئین ناشناخته با مقایسه توالی آن با تاخوردگیهای شناختهشده در PDB پیشبینی میشود. اخیراً، روشهای یادگیری عمیق مانند AlphaFold2 و RoseTTAFold با آموزش روی ساختارهای بانک اطلاعاتی پروتئین، انقلابی در پیشبینی ساختار پروتئین ایجاد کردهاند که دقت پیشبینی را به سطح تجربی نزدیک میکند.
PDB منبعی حیاتی برای طراحی دارو مبتنی بر ساختار (SBDD) است. با دسترسی به ساختار سهبعدی پروتئینهای هدف، محققان میتوانند جایگاههای اتصال برای مولکولهای کوچک را شناسایی کنند و مولکولهای دارویی را طراحی کنند که به طور خاص با این جایگاهها تعامل داشته باشند. روشهای مختلفی مانند داکینگ مولکولی، غربالگری مجازی و طراحی دارو به کمک کامپیوتر بهشدت به ساختارهای بانک اطلاعاتی پروتئین وابسته هستند. داروهای موفق متعددی مانند مهارکنندههای نوروآمینیداز برای درمان آنفولانزا و مهارکنندههای پروتئاز HIV با استفاده از این روشها توسعه یافتهاند. ساختارهای کمپلکس پروتئین-لیگاند در بانک اطلاعاتی پروتئین همچنین اطلاعات ارزشمندی درباره تعاملات مولکولی و مکانیسمهای اثر داروها فراهم میکنند.
ساختارهای بانک اطلاعاتی پروتئین برای مطالعات تکاملی و عملکردی پروتئینها نیز کاربرد گستردهای دارند. مقایسه ساختارهای همولوگ از گونههای مختلف میتواند بینشهای ارزشمندی درباره تکامل عملکرد پروتئین و سازگاری ساختاری فراهم کند. تحلیل ساختاری میتواند جایگاههای حفاظتشده را که معمولاً برای عملکرد پروتئین ضروری هستند، شناسایی کند. همچنین، ساختارهای بانک اطلاعاتی پروتئین برای پیشبینی عملکرد پروتئینهای با عملکرد ناشناخته استفاده میشوند. روشهایی مانند تطبیق ساختاری و شناسایی موتیفهای ساختاری میتوانند ارتباطات عملکردی را بین پروتئینهایی نشان دهند که در سطح توالی شباهت کمی دارند اما دارای تاخوردگیهای مشابه هستند.
در سالهای اخیر، شاهد رشدی تصاعدی در تعداد ساختارهای ذخیرهشده در بانک اطلاعاتی پروتئین هستیم. این رشد به دلیل پیشرفتهای تکنیکی در روشهای تعیین ساختار، به ویژه میکروسکوپ کرایوالکترونی (Cryo-EM) است که امکان تعیین ساختار کمپلکسهای پروتئینی بزرگ و غشایی را فراهم کرده است. همزمان، کیفیت ساختارهای ارائهشده نیز بهبود یافته است. wwPDB استانداردهای اعتبارسنجی سختگیرانهتری را اجرا کرده و ابزارهای اعتبارسنجی پیشرفتهتری را نیز توسعه داده است. روندهای نوظهور مانند خودکارسازی جمعآوری و تحلیل دادهها در کریستالوگرافی، و پیشرفت در پردازش تصویر Cryo-EM، رشد مداوم PDB را تضمین میکنند. چالش آینده، مدیریت و استفاده موثر از این حجم عظیم دادههای ساختاری خواهد بود.
هوش مصنوعی و یادگیری ماشین تأثیر عمیقی بر تحلیل دادههای بانک اطلاعاتی پروتئین گذاشتهاند. الگوریتمهای یادگیری عمیق مانند شبکههای عصبی کانولوشنی (CNNs) و شبکههای عصبی بازگشتی (RNNs) برای پیشبینی تعاملات پروتئین-لیگاند، طبقهبندی پروتئینها بر اساس ساختار و پیشبینی عملکرد از ساختار استفاده میشوند. موفقیت AlphaFold2 در CASP14 نقطه عطفی در پیشبینی ساختار پروتئین بود که با آموزش روی ساختارهای بانک اطلاعاتی پروتئین به دست آمد. روشهای یادگیری ماشین همچنین برای بهبود کیفیت ساختارهای تجربی، مانند بهبود نقشههای تراکم الکترونی و مدلسازی خودکار استفاده میشوند. در آینده، انتظار میرود الگوریتمهای هوش مصنوعی نقش مهمتری در استخراج دانش بیولوژیکی از دادههای PDB و پیشبینی اثرات جهشها و طراحی هدفمند پروتئینها داشته باشند.
یک روند مهم در توسعه بانک اطلاعاتی پروتئین، ادغام آن با سایر دیتابیسهای زیستی است. رویکردهای بیوانفورماتیک مدرن به ترکیب اطلاعات از منابع متعدد نیاز دارند. پروژههایی مانند PDBe-KB (مرکز دانش بانک اطلاعاتی پروتئین اروپا) به دنبال یکپارچهسازی دادههای ساختاری با اطلاعات عملکردی، تکاملی و ژنومیکی هستند. پیوندهایی بین PDB و پایگاههای دادهای مانند UniProt (توالیهای پروتئینی)، KEGG (مسیرهای متابولیکی)، DrugBank (اطلاعات دارویی) و OMIM (بیماریهای ژنتیکی) ایجاد شدهاند. این ادغام امکان تحلیلهای جامعتر را فراهم میکند که میتواند به درک بهتر ارتباط بین ساختار، عملکرد و بیماری کمک کند. آینده PDB احتمالاً شامل افزایش تعاملپذیری با سامانههای بیوانفورماتیک و دیتابیسهای متنوع خواهد بود.
پایگاه PDB و فرمت فایل آن، نقشی اساسی در پیشرفت زیستشناسی ساختاری و توسعه داروهای جدید داشتهاند. از زمان تأسیس در سال 1971، بانک اطلاعاتی پروتئین به یک زیرساخت علمی حیاتی تبدیل شده است که دسترسی آزاد به دادههای ساختاری ماکرومولکولهای زیستی را برای جامعه جهانی فراهم میکند. فرمت استاندارد فایل PDB، به عنوان یک زبان مشترک برای تبادل اطلاعات ساختاری، به توسعه نرمافزارها و روشهای تحلیلی متنوع کمک کرده است. با پیشرفت فناوریهای تعیین ساختار و روشهای محاسباتی، بانک اطلاعاتی پروتئین همچنان به تکامل خود ادامه میدهد تا نیازهای در حال تغییر جامعه علمی را برآورده سازد.
اهمیت بانک اطلاعاتی پروتئین در بیوانفورماتیک را میتوان در تأثیر آن بر کشف دارو، بیوتکنولوژی، زیستشناسی سامانهای و پزشکی شخصیسازیشده مشاهده کرد. با استفاده از ساختارهای بانک اطلاعاتی پروتئین، محققان توانستهاند بینشهای ارزشمندی درباره مکانیسمهای مولکولی بیماریها به دست آورند و درمانهای هدفمند جدیدی توسعه دهند. در آینده، انتظار میرود که PDB با ادغام روشهای هوش مصنوعی، دادههای حجیم و رویکردهای میانرشتهای، نقش مهمتری در پیشبرد دانش و نوآوری در علوم زیستی داشته باشد.
پیشنهاد: آموزش T-Coffee چیست؟ معرفی و آموزش T-Coffee
پاسخ:
PDB (بانک اطلاعاتی پروتئین (Protein Data Bank)) یک دیتابیس برای ذخیرهسازی دادههای سهبعدی ساختاری پروتئینها، اسیدهای نوکلئیک و کمپلکسهای ماکرومولکولی است. این دادهها از روشهای تجربی مانند کریستالوگرافی پرتو ایکس، طیفسنجی NMR و میکروسکوپ کرایوالکترونی (Cryo-EM) به دست میآیند و برای تحقیقات زیستشناسی ساختاری، طراحی دارو و طراحی واکسن استفاده میشوند.
فرمت فایل PDB یک استاندارد متنی است که اطلاعات مربوط به ساختار سهبعدی ماکرومولکولها را ذخیره میکند. این فایل شامل مختصات اتمی، توالی آمینواسیدی، زنجیرههای مولکولی و اطلاعات تکمیلی درباره ساختار است. فایلهای PDB توسط اکثر نرمافزارهای بیوانفورماتیک پشتیبانی میشوند.
برای استفاده از بانک اطلاعاتی پروتئین، میتوانید به وبسایتهای مرتبط مانند RCSB PDB، PDBe یا PDBj مراجعه کنید. از این طریق میتوانید ساختارها را جستجو کنید، دادهها را دانلود کنید و از ابزارهای آنلاین برای تجزیه و تحلیل ساختارها استفاده کنید. همچنین امکان دسترسی برنامهنویسی از طریق API نیز وجود دارد.
برای تفسیر فایلهای PDB، باید رکوردهای کلیدی مانند ATOM (مختصات اتمها)، HETATM (مولکولهای کوچک)، SEQRES (توالی آمینواسیدی) و REMARK (اطلاعات تکمیلی) را بررسی کنید. نرمافزارهایی مانند PyMOL، UCSF Chimera و VMD میتوانند این دادهها را به صورت سهبعدی تجسم کنند و تحلیلهای ساختاری ارائه دهند.
برای ارسال یک ساختار به PDB، باید فایل ساختار را در فرمت مناسب طبق راهنمای موجود در بانک اطلاعاتی پروتئین تهیه کنید، دادههای تجربی را ارائه دهید و فرم مربوطه را تکمیل کنید. این فرآیند شامل بررسی کیفیت و اعتبارسنجی دادهها است تا مطمئن شوند که استانداردهای PDB رعایت شدهاند.
پیشنهاد: آموزش بوت کمپ بیوانفورماتیک چیست؟

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
سلام، وقت بخیر.
چطور میتونیم بهتون کمک کنیم؟ 🤓
تیم ما آماده پاسخگویی به سوالات شماست.
پشتیبانی 24 ساعته در 7 روز هفته.