دوره NGS منتشر شد …

توالی یابی RNA (RNA-Seq) تحول بزرگی در مطالعه انواع مولکول های RNA (transcriptome) در سلولها ایجاد کرده است. این نوع توالی یابی، ابزاری بسیار حساس و دقیق برای سنجش بیان ژن در سطح رونویسی (transcription) بوده، و به محققان امکان کشف تغییراتی را میدهد که در بیماری، پاسخ به درمان، شرایط محیطی مختلف، و طیف وسیعی از طرحهای مطالعاتی دیگر، ناشناخته بوده اند.
RNA-Seq به محققان اجازه میدهد تا هم ویژگیهای شناختهشده و هم جدید را در یک سنجش واحد شناسایی کنند، که امکان تشخیص ایزوفرمهای رونوشت (transcript)، فیوژن های ژنی، واریانت های تک نوکلئوتیدی (SNP) و … را بدون محدودیت دانش قبلی فراهم میکند.

همانطور که مشخص است، داده های RNA-Seq از خوانش های (read) با طول کوتاهmRNA استفاده می کنند که عاری از DNA غیر کد کننده اینترونیک می باشند. این خوانش ها، که در پایان کار به تعداد میلیون ها خوانش می رسد، میتوانند در صورت موجود بودن ژنوم رفرنس، با آن هم ردیف شده یا در صورت عدم وجود ژنوم رفرنس، از نو (de novo) مونتاژ شوند تا یک نقشه توالی RNA تولید کنند که رونوشت را پوشش میدهد.
پیشنهاد: میزکار طراحی پرایمر وانیار امکان طراحی انواع مختلف پرایمرها را به سادگی و با چند کلیک فراهم کرده است. این پرایمرها تحت نظارت کارشناسان بیوانفورماتیک طراحی و اعتبارسنجی می شوند. برای مشاهد پلن های کاربری به صفحه طراحی پرایمر و پروب وارد شوید: ورود به صفحه طراحی پرایمر و پروب.
RNA-Seq با توالی یابی نسل بعدی (NGS) به طور فزاینده ای روش انتخابی برای محققانی است که رونوشت را مطالعه می کنند. این ابزار مزایای بی شماری نسبت به آرایه های بیان ژن (gene expression arrays) ارائه می دهد.
از جمله برتری های RNA-Seq میتوان به سیگنال پسزمینه پایین اشاره کرد. توالیهای cDNA مورد استفاده در RNA-Seq را میتوان به مناطق هدف روی ژنوم مَپ (map) کرد، که حذف نویز آزمایش را آسان میکند.
علاوه بر این، مسائل مربوط به کراس-هیبریداسیون یا هیبریداسیون غیر استاندارد، که میتواند آزمایشهای ریزآرایه (microarray) را دچار اختلال کند، در آزمایشهای RNA-Seq مطرح نیست. به علاوه، داده های ریزآرایه فقط به صورت مقادیری نسبت به سایر سیگنال های شناسایی شده در آرایه نمایش داده می شوند، در حالی که داده های RNA-Seq قابل اندازه گیری هستند. همچنین، RNA-Seq فاقد مشکلات ریزآرایه ها در تشخیص سطوح رونویسی بسیار بالا یا بسیار پایین می باشد.
پیشنهاد: میزکار آنالیز نتایج توالی یابی سنگر امکان آنالیز توالی های مختلف را به سادگی و با چندکلیک فراهم کرده است. نتایج توسط کارشناسان بیوانفورماتیک وانیار گزارش و تایید می شوند. برای مشاهد پلن های کاربری به صفحه آنالیز نتایج توالی یابی سنگر وارد شوید: ورود به صفحه آنالیز نتایج توالی یابی سنگر.
پس از استخراج RNA و کنترل کیفیت آن، نوبت به آماده سازی کتابخانه های RNA-Seq می رسد. انواع واکنش مربوط به مراحل آماده سازی میتواند بسته به روش ساخت کتابخانه مورد استفاده و نوع RNAمورد مطالعه متفاوت باشد، با این حال اصول مولکولی زیربنایی این مراحل یکسان هستند.
واکنشهای زیر معمولاً در تهیه کتابخانه RNA-Seq استفاده میشوند:
رونویسی معکوس (reverse transcription) یا سنتز رشته اول، که به تولید یک مولکول DNA مکمل از روی الگوی RNA، توسط آنزیمی به نامReverse Transcriptase اشاره دارد. سنتز رشته اول میتواند از طریق رونویسی معکوس با پرایمر حاوی اولیگو-dT (پرایمر به طور خاص به دم پلی-A در انتهای 3’ از RNA وصل می شود) یا رونویسی معکوس تصادفی (پرایمرهای تصادفی در امتداد الگوی RNA هیبرید می شوند) انجام شود.
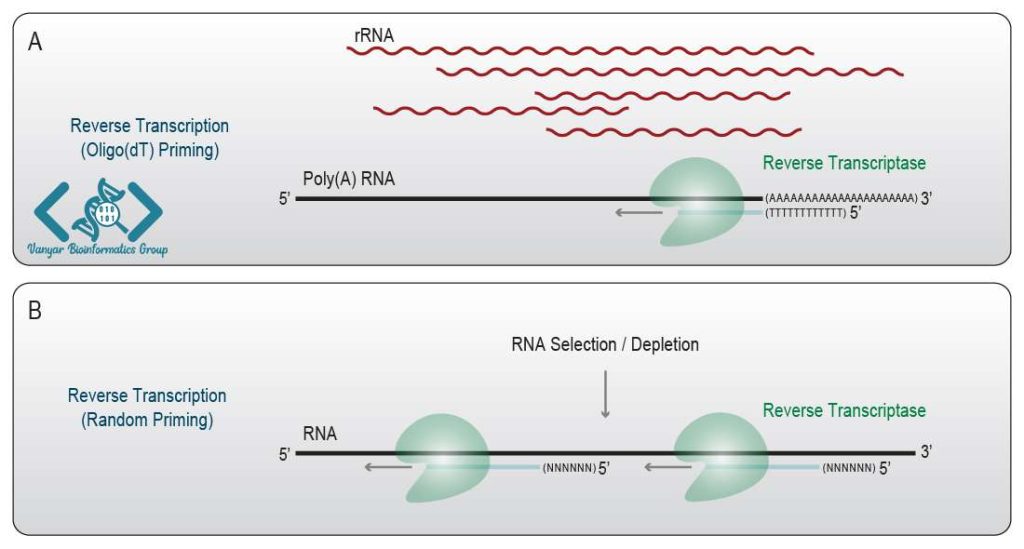
حذف الگوی RNA و سنتز رشته دوم (اضافه شدن نوکلئوتیدهای مکمل با استفاده از DNA پلیمراز)
سنتز رشته اول و دوم، سبب ایجاد DNA دو رشته ای با انتهای تک رشته می شود. بنابراین اغلب برای آماده سازیDNA دو رشته ای جهت اتصال آداپتور، فرآیندی به نام تعمیر انتهایی به کار می رود (شکل پایین). یک مخلوط آنزیمی معمولی ممکن است، برای مثال، حاوی DNA پلیمراز T4 و پلی نوکلئوتید کیناز T4 (PNK) باشد. DNA پلیمراز T4 در حضور dNTP می تواند انتهای آزاد سر 5’ را پر کرده و انتهای آزاد سر 3’ را تا کوتاه کند تا انتهای صاف ایجاد شود.
سپس T4 PNK می تواند نوکلئوتید انتهای 5’ را فسفریله کند. مرحله ی بعد، A-tailing نیز به پلیمراز نیاز دارد. Taq DNA پلیمراز رایج ترین است زیرا دارای فعالیت ترانسفراز انتهایی است و به طور طبیعی یک آدنین انتهایی 3’ بر جای می گذارد.
استفاده از هر یک این دست پلیمرازها انتهای A-tailed را ایجاد می کند که مکمل آداپتورهای توالی استاندارد هستند. افزودن آداپتور در این مرحله فقط نیاز به انکوباسیون با T4 DNA لیگاز دارد. این آنزیم هر دو انتهای صاف و به اصطلاح «چسبنده» را به هم میپیوندد.
همچنین انواع دیگری از “تعمیر انتهایی” در آماده سازی کتابخانه های RNA-Seq استفاده می شود. موضوع اصلی این است که انتها های آزاد برداشته یا پر می شوند. این کار بیشتر روی DNA انجام می شود، اما RNA نیز می تواند تحت تعمیر نهایی قرار گیرد.

PCR آخرین مرحله در آماده سازی اکثر کتابخانه های NGS است. در طی این مرحله، کتابخانه ها برای کنترل کیفیت و در نهایت توالی یابی، تکثیر می شوند.

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
سلام، وقت بخیر.
چطور میتونیم بهتون کمک کنیم؟ 🤓
تیم ما آماده پاسخگویی به سوالات شماست.
پشتیبانی 24 ساعته در 7 روز هفته.